我们知道蛋白质是由不同类型的氨基酸串在一起形成的,氨基酸的组合方式不同,形成的蛋白质在功能上也有所不同。可以计算一下,100个氨基酸能组成的蛋白质结构有10130个,这些结构中只有极少数具有功能性(比例大约为1077分之一)[1]。这给蛋白质的人工合成带来了巨大压力,因为废品率太高了。
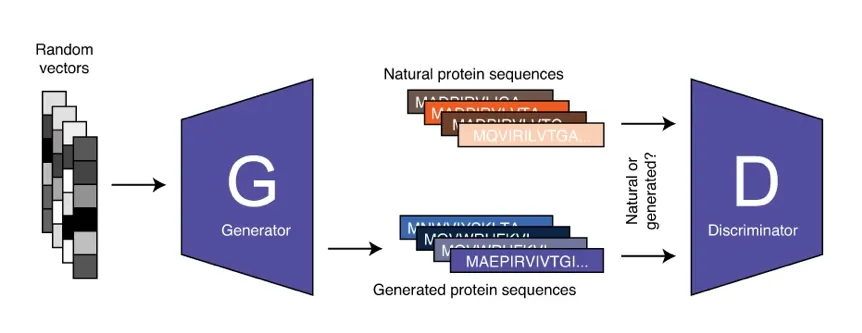
近日Nature 杂志发表了一篇文章,介绍了一种利用深度学习技术合成蛋白质的新方法。他们采用了一种称为对抗生成网络(GAN)的深度学习模型来预测具有功能性的蛋白质,如图1所示。这一模型从一个随机向量开始,通过一个生成器G将其转换成一个蛋白质,再用一个判别器D来检查这个蛋白质和天然蛋白质是否相似,如果不相似的话,就告诉生成器G更新其生成模型。经过学习以后,这一模型就可以生成和天然蛋白质相似的蛋白质了。
图1:基于对抗生成网络的蛋白质生成模型[1]
对抗生成网络已经被广泛应用于各种生成任务中,图2展示了用这种网络生成宠物狗的一个例子[2]。这一模型的基本原理是发现真实数据集中分布的区域,在这一区域随机生成的数据更真实。
图2:基于GAN的宠物狗生成结果[2]

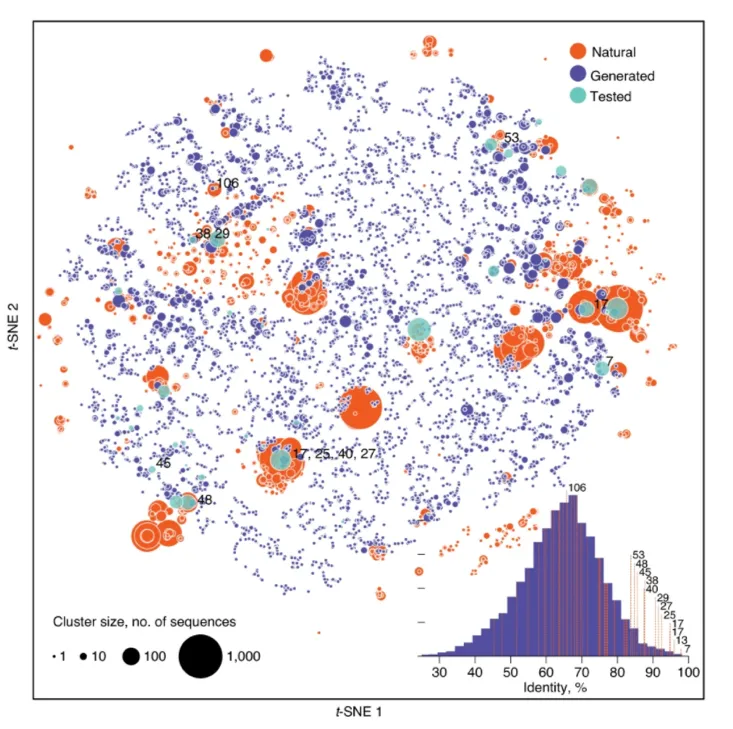
前述蛋白质合成研究正是利用了这一原理,从大量可能性中定位到那些具有功能性的蛋白质结构,实现了更自然的生成。如图3所示,天然蛋白质(橙色圈)分布在对抗生成网络的合成空间中,具有构能性的合成蛋白质(蓝色圈)散布在这些天然蛋白质之间。研究人员利用苹果酸酶(malate dehydrogenase)进行了实验,发现在模型生成的55种蛋白质中,有13种具有催化活性,极大提高了合成效率。
图3:天然蛋白质(橙色圈)与生成的蛋白质(蓝色圈)在空间中的分布[2]
参考文献:
[1]Repecka, D., Jauniskis, V., Karpus, L. et al. Expanding functional protein sequence spaces using generative adversarial networks. Nat Mach Intell (2021).
[2]Yan Wu et al., LOGAN: LATENTOPTIMISATION FORGENERATIVEADVERSARIALNETWORKS, 2020.
